Prédiction d’énergies libres d’hydratation et coefficients de partition solvent/eau par des simulations de dynamique moléculaire
Nous avons développé, en collaboration avec Oliver Beckstein (Arizona State University, Etats-Unis), un ensemble de trois outils afin de faciliter le calcul d’énergies libres de solvatation (ΔGsolv) et de coefficients de partition solvent/eau (logKSW) par des simulations de dynamique moléculaire :
- MDPOW – calcul d’énergies libres de solvatation (ΔGsolv) et de coefficients de partition solvent/eau (logKSW) ;
- MOL2FF – paramétrisation automatisée de ligands dans le champ de force OPLS-AA ;
- Ligandbook – dépôt en ligne pour les topologies et les paramètres de champ de force des ligands.
Plus généralement, ces outils apportent une aide essentielle dans la mise en place des simulations de dynamique moléculaire impliquant des complexes protéine-ligand.

Représentation des trois outils développés dans l’équipe (MDPOW, MOL2FF et Ligandbook) en lien avec les simulations de dynamique moléculaire.
Participation à des compétitions internationales de prédiction SAMPL
En utilisant le logiciel MDPOW et les paramètres de champ de force OPLS-AA obtenus à l’aide du MOL2FF, nous avons participé aux compétitions internationales de prédiction SAMPL3 (2011), SAMPL4 (2013) et SAMPL5 (2015) en obtenant généralement des très bons résultats.
SAMPL3 (2011)
En utilisant ce protocole, nous avons participé en 2011 à la compétition internationale de prédiction SAMPL3 qui demandait de prédire les valeurs de ΔGhyd pour 36 molécules organisées en trois séries, dérivées d’éthane, de biphényle et de dibenzo-p-dioxine, portant un nombre variable d’atomes de chlore. Le facteur déterminant qui nous a départagé par rapport aux autres participants a été de ne pas utiliser seulement les paramètres OPLS-AA disponibles, qui n’étaient pas très adaptés pour les molécules portant un nombre important de substituents chlorés, mais aussi de développer des nouveaux paramètres, avec des charges obtenues par des calculs quantiques avec deux méthodes différentes. Nous avons ainsi pu développer un nouveau set de paramètres transférables pour les molécules portant plus de trois substituents chlorés sur un noyau aromatique.
Dans ces conditions nous avons pu prédire les valeurs de ΔGhyd pour ces composés difficiles avec une « root mean square error » (RMSE) de 1.01 kcal/mol (à comparer avec une RMSE de 4.27 kcal/mol obtenue avec les paramètres OPLS-AA d’origine) et une des nos solutions a été classée première dans la section « Prédiction de l’énergie libre d’hydratation ».
SAMPL4 (2013)
Un protocole similaire a été utilisé en 2013, pour la prédiction d’énergie libre d’hydratation pour 52 composés avec des structures très diverses, dans le cadre de la compétition SAMPL4. Nous avons pu paramétriser deux nouvelles fonctions chimiques, N-alkyl-imidazole et nitrate, et mettre en évidence dans certains cas des problèmes de reproductibilité du terme Lennard-Jones de l’énergie libre, qui sont principalement liés à la flexibilité conformationnelle de la molécule et à la conformation de départ. Globalement, notre prédiction a été réalisée avec une précision (RMSE par rapport aux valeurs expérimentales) tout à fait acceptable de 1,68 kcal/mol.
SAMPL5 (2015)
Dans le cadre de la compétition SAMPL5 nous avons prédit les coefficients de distribution cyclohexane/eau logKCW pour 53 molécules organiques, en utilisant le logiciel MDPOW et les paramètres de champ de force OPLS-AA générés par MOL2FF. Ce travail nous a permis également de valider les paramètres de champ de force OPLS-AA pour le cyclohexane, une condition sine qua non pour réaliser des prédictions de qualité. L’erreur-type (RMSE) de nos prédictions a été de presque 4 unités de log, tandis que l’erreur moyenne (ME) a été de –3 par rapport aux valeurs expérimentales. Ces valeurs, qui sont similaires à celles obtenues par les autres participants, sont moins bonnes que celles attendues par les organisateurs et mettent en évidence les difficultés dans la prédiction des coefficients de distribution solvent/eau. L’erreur moyenne que nous avons obtenu, qui est très éloignée de la valeur nulle attendue, suggère la présence d’une erreur systématique dans nos prédictions, mais seulement les valeurs expérimentales de logKCW ne permettent pas d’identifier précisément la source de cette erreur. Des résultats préliminaires que nous avons obtenus récemment suggèrent que cette erreur provient de l’énergie libre d’hydratation.
D’une manière générale, ces participations aux compétitions SAMPL nous ont permis de paramétriser plusieurs nouvelles fonctions chimiques et de mettre en évidence un certain nombre de problèmes liés au protocole et à l’utilisation du champ de force OPLS-AA, notamment une erreur systématique dans le calcul de l’énergie libre d’hydratation qui est actuellement en cours d’investigation.
Articles :
- Kenney, I. M. ; Beckstein, O. ; Iorga, B. I., Prediction of cyclohexane-water distribution coefficients for the SAMPL5 data set using molecular dynamics simulations with the OPLS-AA force field. J. Comput. Aided Mol. Des. 2016, 30, 1045-1058 [Version en ligne].
- Beckstein, O. ; Fourrier, A. ; Iorga, B. I., Prediction of hydration free energies for the SAMPL4 diverse set of compounds using molecular dynamics simulations with the OPLS-AA force field. J. Comput. Aided Mol. Des. 2014, 28, 265-276 [Version en ligne].
- Beckstein, O. ; Iorga, B. I., Prediction of hydration free energies for aliphatic and aromatic chloro derivatives using molecular dynamics simulations with the OPLS-AA force field. J. Comput. Aided Mol. Des. 2012, 26, 635-645 [Version en ligne].
Actes de congrès :
- Beckstein, O. ; Iorga, B. I. Prediction of hydration free energies for aliphatic and aromatic chloro derivatives using molecular dynamics simulations with the OPLS-AA force field, 243rd ACS National Meeting & Exposition Abstracts of Papers, San Diego, CA, United States, March 25-29 ; American Chemical Society : San Diego, CA, United States, 2012 ; pp COMP-76.
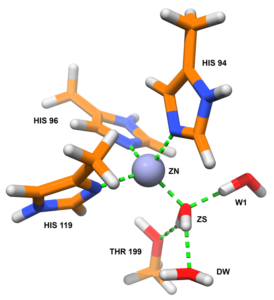
Paramétrisation des sites actifs de métallo-enzymes dans le champ de force OPLS-AA
En utilisant des calculs quantiques et un protocole développé dans l’équipe, nous avons réalisé la paramétrisation des résidus composant le site actif de l’anhydrase carbonique. Les difficultés principales ont été la présence de l’ion de zinc et les deux états de protonation de la molécule d’eau ZS. Ces paramètres ont été ensuite validés par des simulations de dynamique moléculaire.
De manière similaire, nous sommes actuellement en train de paramétriser les sites actifs des 3 sous-classes (B1, B2 et B3) de métallo-β-lactamases qui comportent un (B2) ou deux (B1 et B3) ions zinc.
Articles :
- Bernadat, G. ; Supuran, C. T. ; Iorga, B. I., Carbonic anhydrase binding site parameterization in OPLS-AA force field. Bioorg. Med. Chem. 2013, 21, 1427-1430 [Version en ligne].
Développement d’un protocole efficace de docking pour le criblage virtuel
SAMPL3 (2011)
Dans le cadre de notre participation à la compétition de prédiction SAMPL3 de 2011 dans la section « Criblage virtuel de fragments », à partir d’une librairie de 500 ligands de type fragment il nous a été demandé de classer ces ligands en fonction de leur affinité pour la trypsine bovine. L’étape clé qui nous a différencié par rapport aux autres participants a été l’optimisation préalable du logiciel et des paramètres de docking par rapport au système à étudier. Ainsi, nous avons extrait à partir de la littérature tous les inhibiteurs de trypsine connus, avec leur activité biologique, et sélectionné ceux de type fragment. D’autre part, nous avons analysé toutes les structures de rayons X de trypsine disponibles, complexées avec des ligands de type fragment. Le docking de ces ligands sur la structure de trypsine bovine avec la meilleure résolution, en utilisant deux logiciels de docking (Gold et Glide) et plusieurs fonctions de score, a permis la sélection du logiciel et de la fonction de score qui conduisaient à la meilleure corrélation avec les données biologiques. D’une manière générale, Gold a donné de très bons résultats, tandis que les résultats de Glide ont été plutôt décevants. Avec ce protocole optimisé, nous avons réalisé de docking des 500 fragments, analysé les interactions à l’intérieur du site et généré des courbes de type AUC ROC. Un autre aspect qui nous a probablement différencié par rapport aux autres participants a été l’utilisation des paramètres de recherche conformationnelle typiques pour le docking (100 %) et non de ceux typiques pour le criblage virtuel (30 %). Même si le temps de calcul a été plus long, la meilleure qualité des résultats obtenus a largement justifié ce choix.
En utilisant le protocole de docking optimisé pour le criblage virtuel de fragments comme décrit ci-dessus, nous avons soumis trois solutions qui ont été classées sur les premières trois places dans la section « Criblage virtuel de fragments ». Une de nos images a été sélectionnée pour illustrer la couverture numéro spécial de J. Comput. Aided Mol. Des. dédié à cette compétition.
SAMPL4 (2013)
Un protocole similaire a été utilisé en 2013 pour la section criblage virtuel de la compétition SAMPL4. Le défi était de prédire l’interaction de 321 molécules avec 3 sites différents de la HIV intégrase. Les principales difficultés rencontrées par tous les participants ont été liées à la grande similarité structurale entre les composés actifs et inactifs, ainsi que la présence de 3 sites. Une des nos prédictions a été classée en deuxième place de cette compétition.
CSAR (2014)
Dans le cadre de notre participation à la compétition CSAR 2014, en utilisant un protocole mis au point dans l’équipe nous avons prédit correctement par docking tous les 22 complexes protéine-ligand demandés dans la Phase 1. Les points clé qui nous ont permis d’obtenir ce très bon résultat sont : i) l’analyse préliminaire des logiciels de docking et des fonctions de score disponibles afin de choisir la combinaison qui est la plus adapté à la protéine cible et ii) des paramètres améliorés pour la recherche conformationnelle pendant le docking, au prix d’un temps de calcul beaucoup plus long, afin de mieux explorer l’espace conformationnel des ligands grands et flexibles. Dans la Phase 2, nous avons proposé des poses de docking avec un RMSD moyen de 1-2 Å, en fonction des caractéristiques de chaque série. Par contre, le classement des poses s’est révélé plus problématique, mettant en lumière les limitations des fonctions de score disponibles actuellement. Une image de ce travail a été sélectionnée pour la couverture du numéro spécial du J. Chem. Inf. Model. dédié à la compétition CSAR. Notre publication a été l’article open access ACS AuthorChoice le plus lu du journal J. Chem. Inf. Model. en 2016.
D3R Grand Challenge (2015)
Dans le cadre de notre participation à la compétition D3R Grand Challenge 2015 il nous a été demandé de réaliser la prédiction de l’affinité relative pour 180 ligands de la protéine Heat Shock Protein 90 (HSP90) et du mode d’interaction pour 6 d’entre eux, ainsi que le mode d’interaction pour 30 ligands de la protéine Mitogen-Activated Protein Kinase Kinase Kinase Kinase 4 (MAP4K4) et la prédiction de l’affinité relative pour 18 d’entre eux. Les ligands HSP90 faisaient partie de trois familles (benzimidazolones, aminopyrimidines, et dérivés de benzophénone), tandis que les ligands MAP4K4 étaient très divers.
Une analyse approfondie des données expérimentales (structurales et biochimiques) disponibles a montré que Gold avec la fonction de score GoldScore donnent les meilleurs résultats avec ces deux protéines, et que les résidus Lys58 (HSP90) et Lys54 (MAP4K4) doivent être flexibles pendant le docking. Dans le cas de HSP90, le docking a été réalisé sur 11 structures représentatives pour les conformations des 191 structures cristallographiques disponibles dans Protein Data Bank (PDB). De cette manière, nous avons mieux exploré l’espace conformationnel de cette protéine qui est très flexible dans la région des résidus 99-129.
La prédiction du mode d’interaction des premiers quatre ligands avec HSP90 a été excellente, à l’exception du groupe pyridylsulfonyle dans HSP044, tandis que pour les deux derniers (HSP175 et HSP179) nous avons globalement bien prédit l’orientation dans le site actif, mais avec des interactions un peu différentes. Ces résultats correspondent à un RMSD moyen de 1,48 Å pour les conformations classées en premier, et à un RMSD moyen de 1,20 Å pour les poses avec le meilleur RMSD. Pour MAP4K4, nous avons prédit correctement 11 ligands qui présentaient un mode d’interaction « classique » et incorrectement 17 ligands qui présentaient un nouveau mode d’interaction et 2 ligands qui interagissaient avec le site actif à travers des molécules d’eau.
Comme pour les compétitions précédentes, la prédiction des affinités relatives des ligands a été plus difficile pour la plupart des participants. Cependant, notre soumission pour MAP4K4 a été classée 2ème parmi 46 soumissions.
Ce travail a mis en évidence la nécessité d’identifier les outils et les paramètres de docking qui sont les plus adaptés à chaque cible, ainsi que les résidus du site actif qui doivent être flexibles pendant le processus de docking. Des traitements supplémentaires des poses de docking, notamment par des calculs d’énergie libre, semblent nécessaires pour améliorer la prédiction de l’affinité relative des ligands, notamment en tenant compte de l’influence des molécules d’eau et de la flexibilité globale de la protéine, et nos efforts futurs seront concentrés dans cette direction.
Articles :
- Selwa, E. ; Martiny, V. Y. ; Iorga, B. I., Molecular docking performance evaluated on the D3R Grand Challenge 2015 drug-like ligand datasets. J. Comput. Aided Mol. Des. 2016, 30, 829-839 [Version en ligne].
- Martiny, V. Y. ; Martz, F. ; Selwa, E. ; Iorga, B. I., Blind pose prediction, scoring, and affinity ranking of the CSAR 2014 dataset. J. Chem. Inf. Model. 2016, 56, 996-1003 [<ahref= »http://dx.doi.org/10.1021/acs.jcim.5b00337″>Version en ligne] (Open Access).
- Colas, C. ; Iorga, B. I., Virtual screening of the SAMPL4 blinded HIV integrase inhibitors dataset. J. Comput. Aided Mol. Des. 2014, 28, 455-462 [<ahref= »http://dx.doi.org/10.1007/s10822-014-9707-5″>Version en ligne].
- Surpateanu, G. ; Iorga, B. I., Evaluation of docking performance in a blinded virtual screening of fragment-like trypsin inhibitors. J. Comput. Aided Mol. Des. 2012, 26, 595-601 [<ahref= »http://dx.doi.org/10.1007/s10822-011-9526-x »>Version en ligne].